Перейти к:
Сравнительный анализ нанопоровых секвенаторов MinION и Нанопорус в задаче идентификации нуклеиновых кислот патогенов
https://doi.org/10.47183/mes.2024-242
Аннотация
Введение. Технологии нанопорового секвенирования стали рутинным инструментом в науке и медицине, широко применяются в исследовании разнообразия и распространения патогенов, играют ключевую роль в полевой эпидемиологии.
Цель. Проведение сравнительного анализа функциональных возможностей секвенаторов третьего поколения MinION и Нанопорус в задаче выявления патогенов в биологическом материале, включая сопоставление таксономического состава, определенного с их использованием, с результатами, полученными на референсной платформе второго поколения MiSeq (Illumina).
Материалы и методы. Проведено исследование 138 образцов архивной ДНК с известным таксономическим составом (исследованы 14 семейств, 20 родов и 43 вида патогенов вирусной и бактериальной природы, суммарно 169 возбудителей инфекций). В исследовании использовались нанопоровые секвенаторы MinION и Нанопорус с оригинальными проточными ячейками R9.4.1 и R10.4.1 от ONT, а также высокопроизводительная платформа MiSeq от Illumina для предварительной идентификации состава исследуемых образцов, содержащих различные титры нуклеиновых кислот возбудителей инфекций ряда таксономических групп. Сравнительный анализ полученных данных (количество последовательностей, средние показатели качества прочтений (Qscore) для каждого нуклеотида, GC-состав последовательностей, распределение длин последовательностей, уровень дупликаций прочтений) проводился биоинформатическим инструментом MultiQC (версия 1.20).
Результаты. В ходе проведенных исследований на приборах MinION и Нанопорус было идентифицировано 98,8 и 97,6% патогенов соответственно, включая малоизученные или новые вирусы. Применение последней версии проточной ячейки на обоих приборах значительно снизило долю низкокачественных прочтений. Полученные данные продемонстрировали высокую степень корреляции между результатами секвенаторов второго и третьего поколений, что подтверждает сопоставимость и взаимозаменяемость этих технологий в задаче идентификации нуклеиновых кислот патогенов.
Выводы. Результаты исследования демонстрируют потенциал нанопоровых секвенаторов MinION и Нанопорус для применения в эпидемиологическом надзоре. Приборы способны обеспечивать высокую точность идентификации патогенов различной природы и благодаря своей компактности и портативности могут существенно повысить скорость диагностики и мониторинга инфекционных заболеваний.
Для цитирования:
Григорян Д.А., Стеценко И.Ф., Гуков Б.С., Мацвай А.Д., Шипулин Г.А. Сравнительный анализ нанопоровых секвенаторов MinION и Нанопорус в задаче идентификации нуклеиновых кислот патогенов. Медицина экстремальных ситуаций. 2025;27(1):64-73. https://doi.org/10.47183/mes.2024-242
For citation:
Grigoryan D.A., Stetsenko I.F., Gukov B.S., Matsvay A.D., Shipulin G.A. Comparative evaluation of MinION and Nanoporus nanopore sequencers in identification of pathogen nucleic acids. Extreme Medicine. 2025;27(1):64-73. https://doi.org/10.47183/mes.2024-242
ВВЕДЕНИЕ
На сегодняшний день технологии секвенирования вошли в список рутинных методов, применяемых во многих областях молекулярной биологии, что позволяет оперативно и достоверно выявлять мутации и генетические вариации вирусов, идентифицировать новые патогены, прогнозировать их эволюционные изменения, отслеживать динамику распространения в популяциях и анализировать филогенетические связи [1–3]. Нанопоровое секвенирование было впервые представлено компанией Oxford Nanopore Technologies (ONT) в 2014 году с устройством MinION [4]. Данная технология обладает множеством уникальных преимуществ, обусловливающих ее востребованность в современной медицине и науке [5]. Благодаря способности секвенировать длинные фрагменты ДНК и РНК стало возможным обнаружение структурных вариаций и эпигенетических модификаций [6–8]. Однако ключевым достоинством данного метода секвенирования является компактность нанопоровых секвенаторов, их способность работать посредством подключения к USB-интерфейсу ноутбука, а также низкие требования к оснащенности лаборатории, что открывает возможности применения данных приборов в различных условиях, включая полевые исследования [9].
Технология нанопорового секвенирования уже оказала значительное влияние на различные области медицины, включая диагностику и лечение генетических заболеваний [10][11], персонализированную медицину [2][12], исследование рака [6][13][14]. Благодаря своей скорости и мобильности нанопоровое секвенирование является значимым инструментом для эпидемиологического надзора и контроля за вспышками заболеваемости [9][15–17]. В частности, в условиях пандемии COVID-19 нанопоровое секвенирование сыграло существенную роль в оперативной идентификации штаммов вирусов и выявлении новых генетических вариаций патогенов [12][17][18]. Также данная технология зарекомендовала себя в качестве надежной альтернативы традиционным методам секвенирования полных геномов вирусов [15][19]. Эти качества особенно значимы в условиях глобальных пандемий и вспышек новых инфекций, поскольку оперативно полученные данные играют решающее значение в процессе принятия решений в области общественного здравоохранения.
Помимо оригинальных платформ третьего поколения от компании Oxford Nanopore Technologies, на рынке присутствует аналогичное по функционалу устройство российского производства — Нанопорус. Данный прибор предназначен для нанопорового секвенирования с использованием оригинальных проточных ячеек от Oxford Nanopore Technologies.
Цель исследования — проведение сравнительного анализа функциональных возможностей секвенаторов третьего поколения MinION и Нанопорус в задаче выявления патогенов в биологическом материале, включая сопоставление таксономического состава, определенного с их использованием, с результатами, полученными на референсной платформе второго поколения MiSeq (Illumina).
МАТЕРИАЛЫ И МЕТОДЫ
В рамках двух этапов исследования было проанализировано 138 образцов архивной ДНК из коллекции лаборатории с известным таксономическим составом и различным титром нуклеиновых кислот возбудителей инфекций ряда таксономических групп (исследованы 14 семейств, 20 родов и 43 вида патогенов вирусной и бактериальной природы, суммарно 169 возбудителей инфекций). Идентификация патогенного состава исследуемого материала была предварительно проведена посредством высокопроизводительного секвенирования на платформе MiSeq (Illumina). На основании данных локального выравнивания нуклеотидных и белковых последовательностей выполняли анализ показателей схожести с последовательностями из базы данных, применяемых для таксономической идентификации.
В ходе подготовки ампликонных ДНК-библиотек применяли следующие наборы реагентов: восстановление концов двуцепочечных фрагментов ДНК и безматричное аденилирование проведено набором реагентов NEBNext Ultra II End Repair/dA-Tailing Module (New England Biolabs), лигирование адаптерной последовательности из состава набора PCR Barcoding Expansion 1-96 (EXP-PBC096) (ONT) проводили с применением реагента Blunt/TA Ligase Master Mix (New England Biolabs), баркодирование библиотек осуществлялось набором баркодов PCR Barcoding Expansion 1-96 (EXP-PBC096) (ONT). Все наборы использовались согласно инструкции производителя.
Подготовку ДНК-библиотек для секвенирования на проточной ячейке R9.4.1 проводили с применением набора реагентов Ligation Sequencing Kit (SQK-LSK109) (ONT) с последующей загрузкой в проточную ячейку. Подготовку ДНК-библиотек для секвенирования на проточной ячейке R10.4.1 проводили набором реагентов Ligation Sequencing Kit V14 (SQK-NBD114) (ONT) с последующей загрузкой в проточную ячейку. Последовательность применения секвенаторов определяли с учетом технических и методологических соображений. На первом этапе исследования, при первичном использовании проточной ячейки R9.4.1, секвенирование сначала проводили на секвенаторе MinION, который обеспечивал заведомо стабильную работу ячейки. Этот порядок был выбран для минимизации потенциальных рисков, связанных с возможным снижением стабильности или функциональности ячейки после ее применения на секвенаторе Нанопорус. На втором этапе с целью тестирования секвенатора Нанопорус на воздействие на функциональные характеристики ячейки для последующего использования в приборе MinION секвенирование проводили сначала на Нанопорусе, а затем на MinION.
Запись детектируемого сигнала с приборов осуществляли программным обеспечением MinKNOW версии 23.11.4; бейсколинг данных в формате pod5 проводили программой Dorado версии 7.2.13 (ONT). С целью обеспечения корректности сравнения качества секвенирования и таксономической идентификации тестируемыми приборами для каждого образца отбирали одинаковое количество прочтений, представляющих случайную выборку, сформированную посредством биоинформатического инструмента SeqKit (версия v2.8.0). Оценку качества прочтений в формате fastq осуществляли с применением биоинформатических инструментов Trimmomatic (версия 0.32), FastQC (версия v0.12.0). Сравнительный анализ полученных данных (количество последовательностей, средние показатели качества прочтений (Qscore) для каждого нуклеотида, GC-состав последовательностей, распределение длин последовательностей, уровень дупликаций прочтений) проводили биоинформатическим инструментом MultiQC (версия 1.20). Таксономическую идентификацию вирусного состава образцов проводили с использованием программного обеспечения PathogenID (ФМБА, Россия). В качестве референсного инструмента для сопоставления результатов определения таксономического состава образцов использовались данные, полученные на платформе второго поколения (MiSeq, Illumina).
Для проведения сравнительного анализа тестируемых секвенаторов исследуемые инфекционные агенты разделяли на 5 групп в соответствии с показателем таксономической идентификации (% Identity), полученным в результате высокопроизводительного секвенирования. Группам вирусов присваивали следующие идентификаторы: Группа № 1 — 30 патогенов, 100–97% Identity, Группа № 2 — 29 патогенов, 96–94 % Identity, Группа № 3 — 39 патогенов, 93–90 % Identity, Группа № 4 — 35 патогенов, 89–80 % Identity, Группа №5 — 24 патогена, 79–71 % Identity. Представленные группы патогенов моделируют анализ дивергентных групп патогенов, в частности новых штаммов, видов.
РЕЗУЛЬТАТЫ ИССЛЕДОВАНИЯ
Сравнение стабильности передачи данных и качества секвенирования
С использованием программного обеспечения MinKNOW выполнена оценка стабильности передачи данных с прибора на управляющий компьютер. В ходе анализа графиков, полученных с приборов MinION и Нанопорус с использованием проточной ячейки R10.4.1, значительных флуктуаций и сбоев в передаче сигнала, а также отклонений от равномерного распределения скорости транслокации ДНК через пору не зарегистрировано (рис. 1 А2, Б2). Графики поддержания температурного режима на протяжении всего процесса секвенирования указывали на отсутствие нарушений в работе секвенаторов, колебания температуры были незначительными и оставались в пределах допустимых значений, соответствующие данные представлены на рисунке 1 А1, Б1.
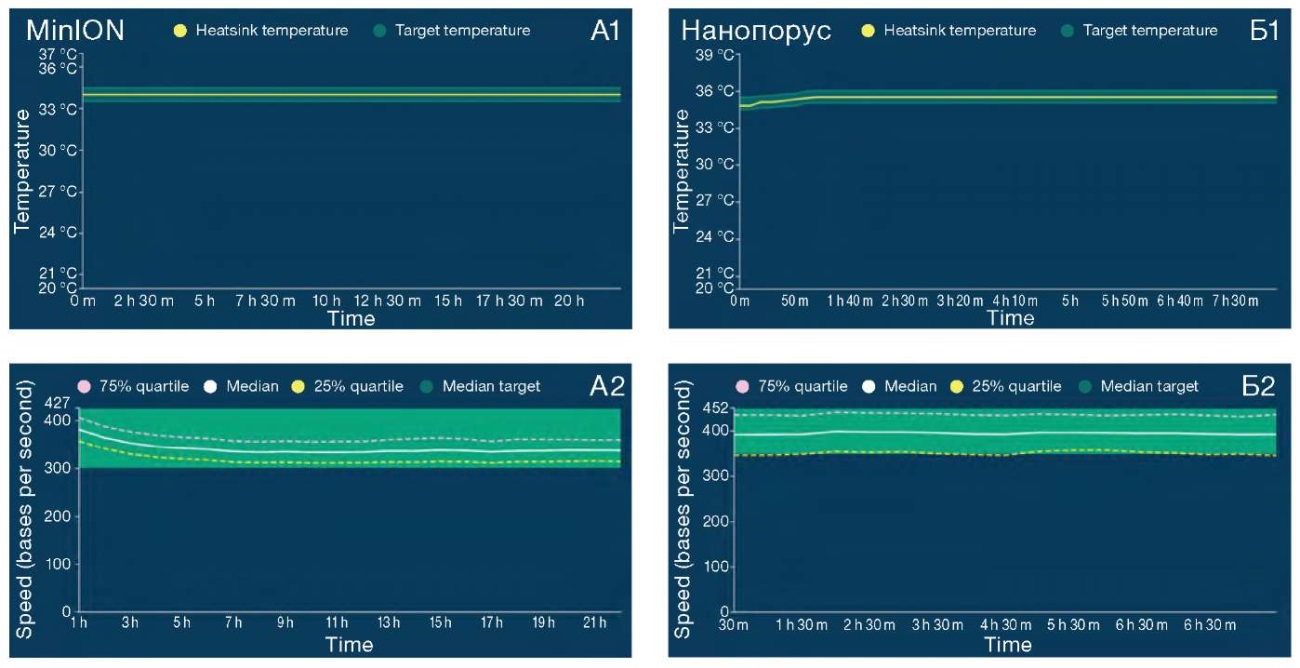
Рисунок подготовлен авторами
Рис. 1. Графическое представление ключевых параметров работы секвенаторов MinION и Нанопорус
Примечание: A1, Б1 — поддержание температурного режима в ходе секвенирования; A2, Б2 — скорость транслокации ДНК через пору.
Графики получены с использованием программного обеспечения MinKNOW версии 23.11.4.
Проведено сравнение показателей качества прочтений, полученных с проточных ячеек как старого, так и нового типа для секвенаторов MinION и Нанопорус (рис. 2). Анализ представленных графиков установил увеличение значения параметра Qscore, отражающего точность идентификации оснований в прочтениях, и снижение уровня дупликаций при использовании проточной ячейки последней версии R10.4.1, также установлено соответствие всех представленных метрик оценки качества прочтений, полученных с двух сравниваемых нанопоровых секвенаторов.
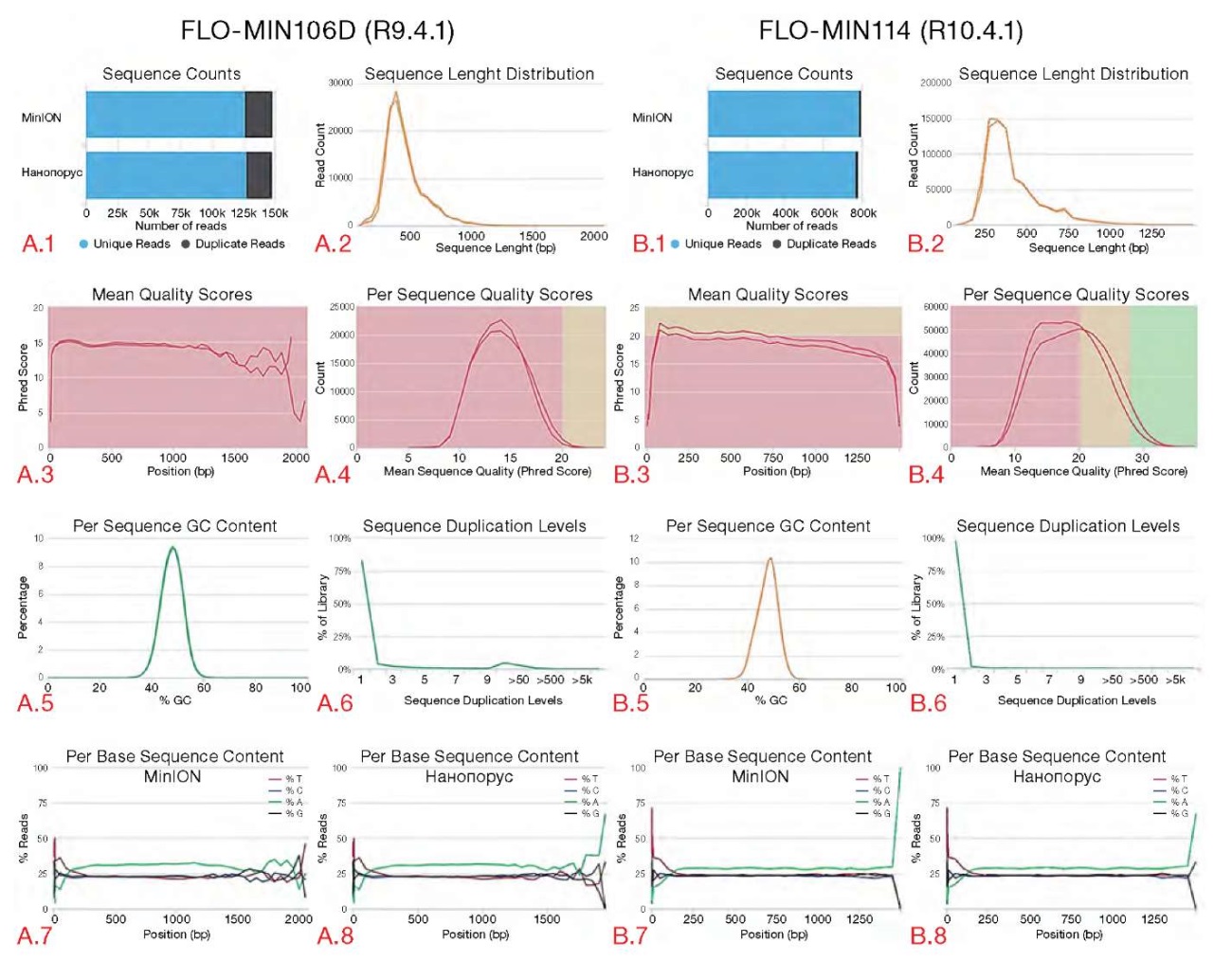
Рисунок подготовлен авторами
Рис. 2. Сравнение показателей качества прочтений, полученных с секвенаторов MinION и Нанопорус
Примечание: график A.1 — общее количество последовательностей, полученных с каждого прибора; график A.2 — распределение ридов по длине прочтений; график A.3 — средние значения качества прочтений (Qscore) для каждого нуклеотида; график A.4 — количество прочтений в зависимости от показателя качества (Qscore); график A.5 — содержание GC в последовательностях, выраженное в процентах; график A.6 — уровень дупликаций прочтений; график A.7 — нуклеотидный состав прочтений, полученных секвенатором MinION; график A.8 — нуклеотидный состав прочтений, полученных секвенатором Нанопорус.
Соответствующие данные для проточной ячейки R10.4.1 показаны на графиках Б.1–Б.8.
Графики соответствуют данным, зарегистрированным при использовании проточных ячеек R9.4.1 и R10.4.1.
Показатели качества прочтений получены биоинформатическим инструментом MultiQC (версия 1.20).
Рассчитаны дополнительные показатели качества секвенирования, а именно метрики N50, N95, N5, а также процент ридов с качеством выше Q20 и Q30, соответствующие данные представлены в таблице 1. В ходе сравнительного анализа процента последовательностей с качеством выше Q20 и Q30 установлено, что при использовании проточной ячейки более ранней версии (R9.4.1) около 70% данных имеют качество ниже Q20 и примерно 97,3% — ниже Q30 на обоих секвенаторах. В то время как при применении проточной ячейки последней версии R10.4.1, доля данных с качеством ниже Q20 составляет менее 55%, а ниже Q30 — менее 76%. Существенных различий в показателях N50, N95, N5, а также в проценте последовательностей с качеством выше Q20 и Q30 между двумя различными приборами третьего поколения не выявлено.
Таблица 1. Показатели качества секвенирования на оригинальных проточных ячейках R10.4.1 и R9.4.1 на секвенаторах третьего поколения Нанопорус и MinION
Параметры оценки качества прочтений | Проточная ячейка R10.4.1, секвенатор MinION | Проточная ячейка R10.4.1, секвенатор Нанопорус | Проточная ячейка R9.4.1, секвенатор MinION | Проточная ячейка R9.4.1, секвенатор Нанопорус |
Средняя длина прочтения, ед. нуклеотидов | 407,8 | 417,1 | 447 | 435 |
Максимальная длина прочтения, ед. нуклеотидов | 1500 | 1500 | 1399 | 1388 |
N50 | 411 | 425 | 454 | 446 |
N5 | 250 | 256 | 289 | 275 |
N95 | 900 | 904 | 871 | 860 |
Таблица подготовлена авторами по собственным данным
Анализ корреляции данных идентификации патогенов между приборами
Исследуемый материал содержал фрагменты геномов патогенов следующих семейств: Pseudomonadaceae, Circoviridae, Adenoviridae, Coronaviridae, Orthomyxoviridae, Parvoviridae, Polyomaviridae, Astroviridae, Caliciviridae, Picornaviridae, Solemoviridae, Hepeviridae, Partitiviridae, Tymoviridae.
Семейство Adenoviridae насчитывало 12 образцов и 5 видов патогенов с вирусной нагрузкой от 7,22 до 0,33% прочтений на образец по данным высокопроизводительного секвенирования. В результате обработки данных, полученных приборами MinION и Нанопорус, таксономический состав был подтвержден у 11 (92%) и 10 (83%) образцов соответственно. Семейство Circoviridae насчитывало 35 образцов и 11 различных видов патогенов с вирусной нагрузкой от 57,27 до 0,04% прочтений по данным секвенирования на платформе Illumina. В результате обработки данных с секвенаторов MinION и Нанопорус таксономический состав был подтвержден у 34 (97%) и 33 (94%) образцов соответственно. Несмотря на то что идентификация целевого патогена нанопоровыми секвенаторами была проведена не для 100% образцов, содержащих вирусы данных семейств (Adenoviridae и Circoviridae), в ходе выравнивания нуклеотидных последовательностей на референсные базы данных для ряда образцов было получено более детальное типирование вирусов нанопоровыми данными.
Согласно данным высокопроизводительного секвенирования на платформе Illumina семейство Coronaviridae насчитывало 70 образцов и 9 различных видов патогенов с вирусной нагрузкой от 49,52 до 0,15% прочтений; выборка образцов семейства Orthomyxoviridae насчитывала 6 образцов и 1 вид с вирусной нагрузкой от 28,97 до 1,32% прочтений; выборка образцов семейства Pseudomonadaceae насчитывала 12 образцов и 1 вид; тестируемая группа семейства Parvoviridae насчитывала 15 образцов и 11 видов патогенов с вирусной нагрузкой от 28,12 до 0,22% прочтений; семейство Picornaviridae насчитывало 4 образца и 4 вида патогенов с вирусной нагрузкой от 11,53 до 0,3% прочтений. И так же, как и у семейств Astroviridae, Caliciviridae, Polyomaviridae, Solemoviridae, Tymoviridae, Partitiviridae и Hepeviridae, насчитывающих единичные образцы, идентификация таксономического состава семейств Coronaviridae, Parvoviridae, Picornaviridae и Orthomyxoviridae проведена у 100% образцов в ходе секвенирования третьего поколения на приборах MinION и Нанопорус. Суммарно нанопоровыми секвенаторами MinION и Нанопорус было идентифицировано 167 (98,8%) и 165 (97,6%) патогенов соответственно.
Для наиболее представленных семейств вирусов проводили сравнение процента прочтений, приходящихся на целевой возбудитель инфекций. Для более доступной визуализации сопоставления данные взяты в логарифмической шкале и проанализированы для трех пар секвенаторов: MiSeq и MinION (A), MiSeq и Нанопорус (Б), а также MinION и Нанопорус (В) (рис. 3). На графиках отмечали возрастание уровня корреляции между результатами секвенирования по мере увеличения вирусной нагрузки в образцах, в особенности между данными нанопорового секвенирования при уровне нагрузки более двух логарифмических единиц. При низкой вирусной нагрузке выявлены отклонения, в частности между данными, полученными на платформе Illumina, и данными секвенаторов третьего поколения.
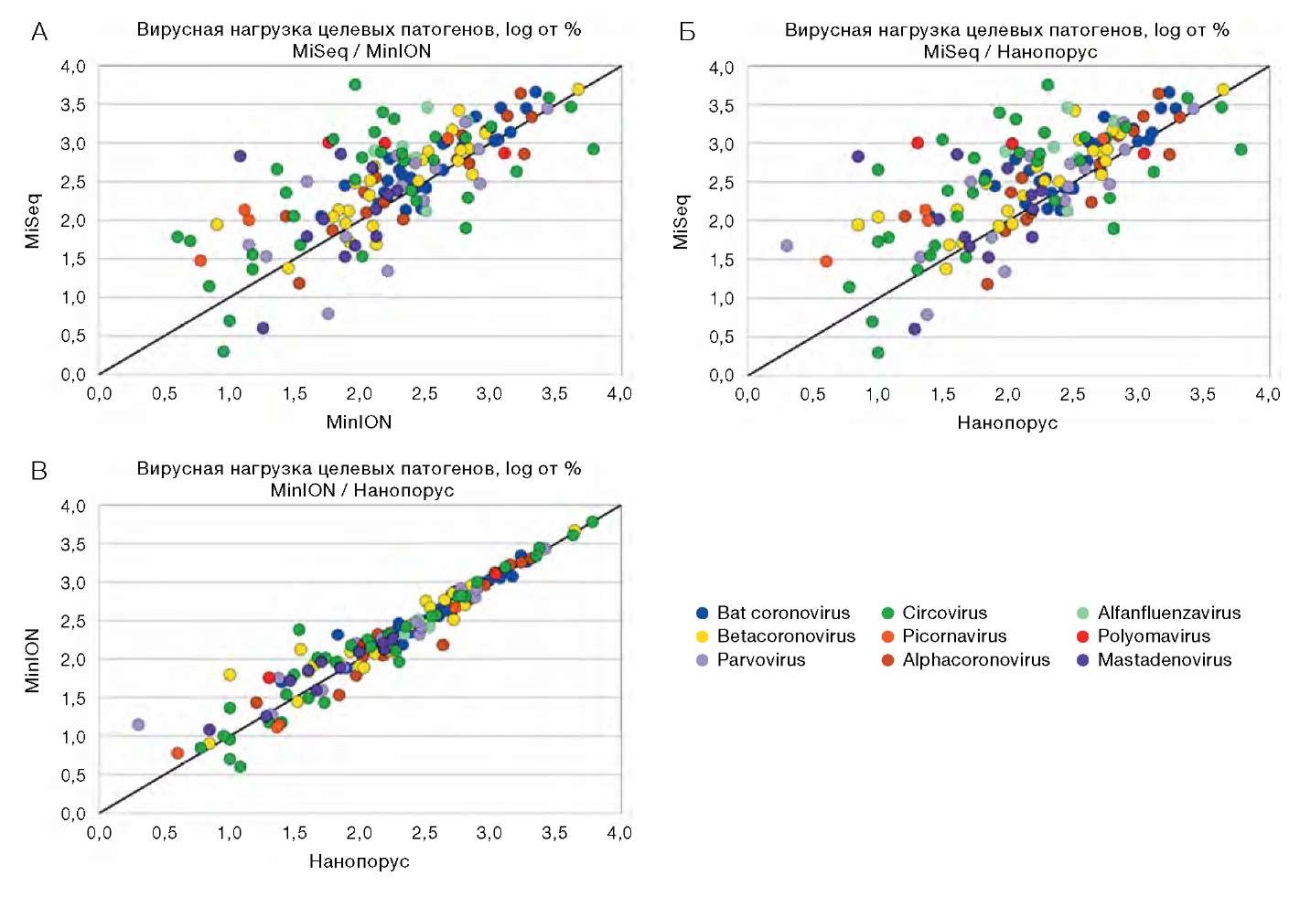
Рисунок подготовлен авторами
Рис. 3. Сравнение процентного содержания прочтений целевых патогенов наиболее представленных семейств
Примечание: график А — секвенаторы MiSeq и MinION; график Б — секвенаторы MiSeq и Нанопорус; график В — секвенаторы MinION и Нанопорус.
Точка на графиках — один исследуемый вирус.
Значения вирусной нагрузки представлены в логарифмической шкале.
Для оценки взаимосвязи показателей процентной вирусной нагрузки на трех приборах рассчитывали коэффициент корреляции Пирсона. Анализ данных, собранных с обеих проточных ячеек, показал следующие результаты: коэффициент корреляции между платформами MiSeq и MinION составлял r = 0,567 (p ≤ 0,05), что указывало на умеренную положительную зависимость. Между MiSeq и Нанопорус зафиксирован коэффициент r = 0,544 (p ≤ 0,05), также свидетельствующий об умеренной положительной связи. Наивысшее значение корреляции наблюдалось между секвенаторами Нанопорус и MinION, равное r = 0,993 (p ≤ 0,05), что указывало на практически полное совпадение результатов между этими двумя приборами.
В ходе исследования сравнивали группы инфекционных агентов, сформированные согласно диапазону таксономической идентичности (% Identity), по средним значениям показателей таксономической идентификации (e-value, % Identity, длина выравнивания (пары нуклеотидных оснований п.о.), процент прочтений целевого вируса), полученным на тестируемых приборах (рис. 4).
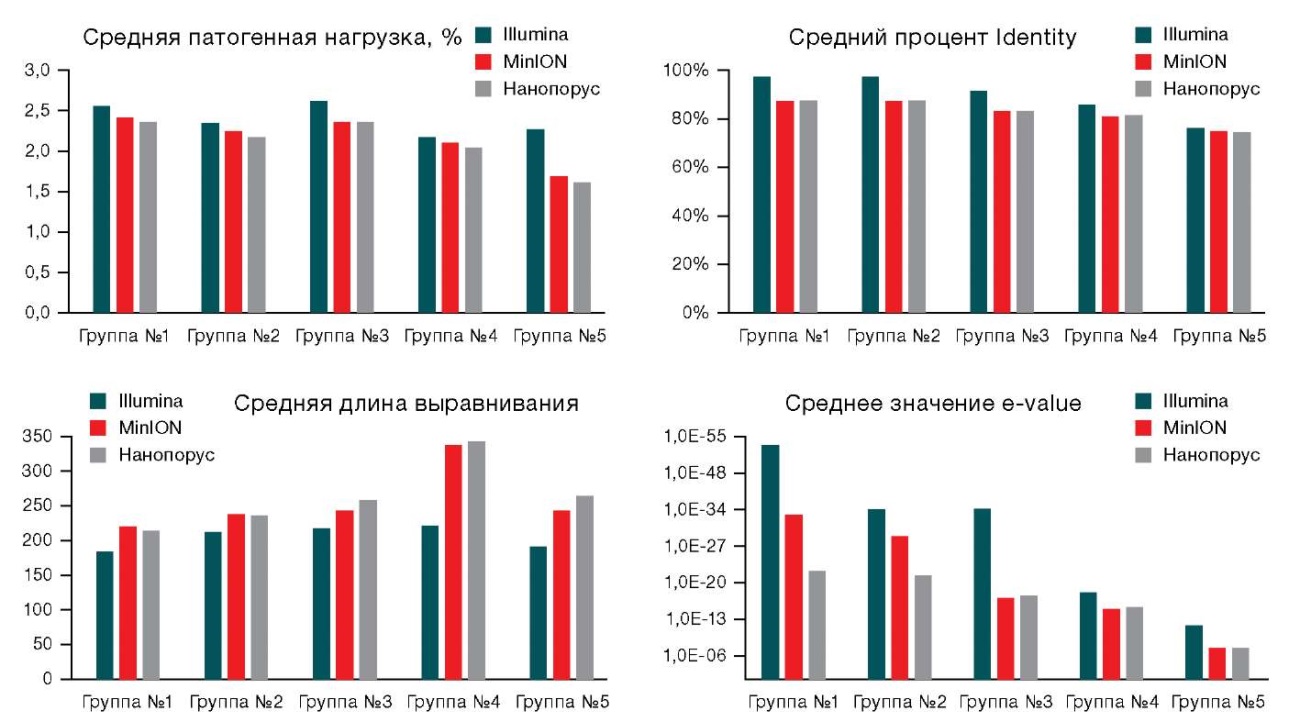
Рисунок подготовлен авторами
Рис. 4. Сравнение средних значений параметров таксономической идентификации (патогенная нагрузка, % Identity, длина выравнивания, e-value), полученных в результате секвенирования на приборах MiSeq, MinION и Нанопорус
Примечание: анализ выполнен на основании данных, полученных с проточных ячеек двух последних версий.
Для всех представленных групп патогенов показатели % Identity и e-value, полученные на платформе MiSeq, незначительно превышали аналогичные показатели, полученные на нанопоровых секвенаторах. В то же время показатель длины выравнивания последовательностей демонстрировали обратную зависимость. Гистограммы указывали на высокую степень соответствия показателей выявления инфекционных агентов между тремя приборами.
Для визуализации различий в достоверности таксономической идентификации, обеспечиваемой тестируемыми секвенаторами, проводили моделирование вариационного распределения вирусной нагрузки и процента идентичности на основании данных, полученных с трех приборов (рис. 5). По результатам первого этапа исследования (95 образцов) (A) наблюдали следующую модель распределения показателей: на платформе MiSeq основное скопление данных располагалось в диапазоне высокой идентичности (90–100%) и средней вирусной нагрузки (2–3 логарифмические единицы); при использовании проточной ячейки R9.4.1 платформа MinION демонстрировала плотное скопление данных в области с высокой идентичностью (более 90%) и относительно высокой вирусной нагрузкой (2–3 логарифмические единицы); данные с секвенатора Нанопорус распределялись шире по оси идентичности, однако основное дисперсионное скопление также находилось в пределах 90% идентичности. По результатам второго этапа исследования (43 образца, предположительно содержащих нуклеиновые кислоты малоизученных патогенов) (Б) получили следующий профиль данных: на платформе MiSeq наблюдалось смещение плотности данных в сторону более низкой идентичности и вирусной нагрузки, что объясняется особенностями исследуемого материала; при использовании проточной ячейки R10.4.1 данные с MinION концентрировались в узком диапазоне 85% идентичности и относительно высокой вирусной нагрузки (2–3 логарифмические единицы); секвенатор Нанопорус демонстрировал менее плотное распределение данных относительно показателей своего аналога, но сопоставимую вирусную нагрузку.
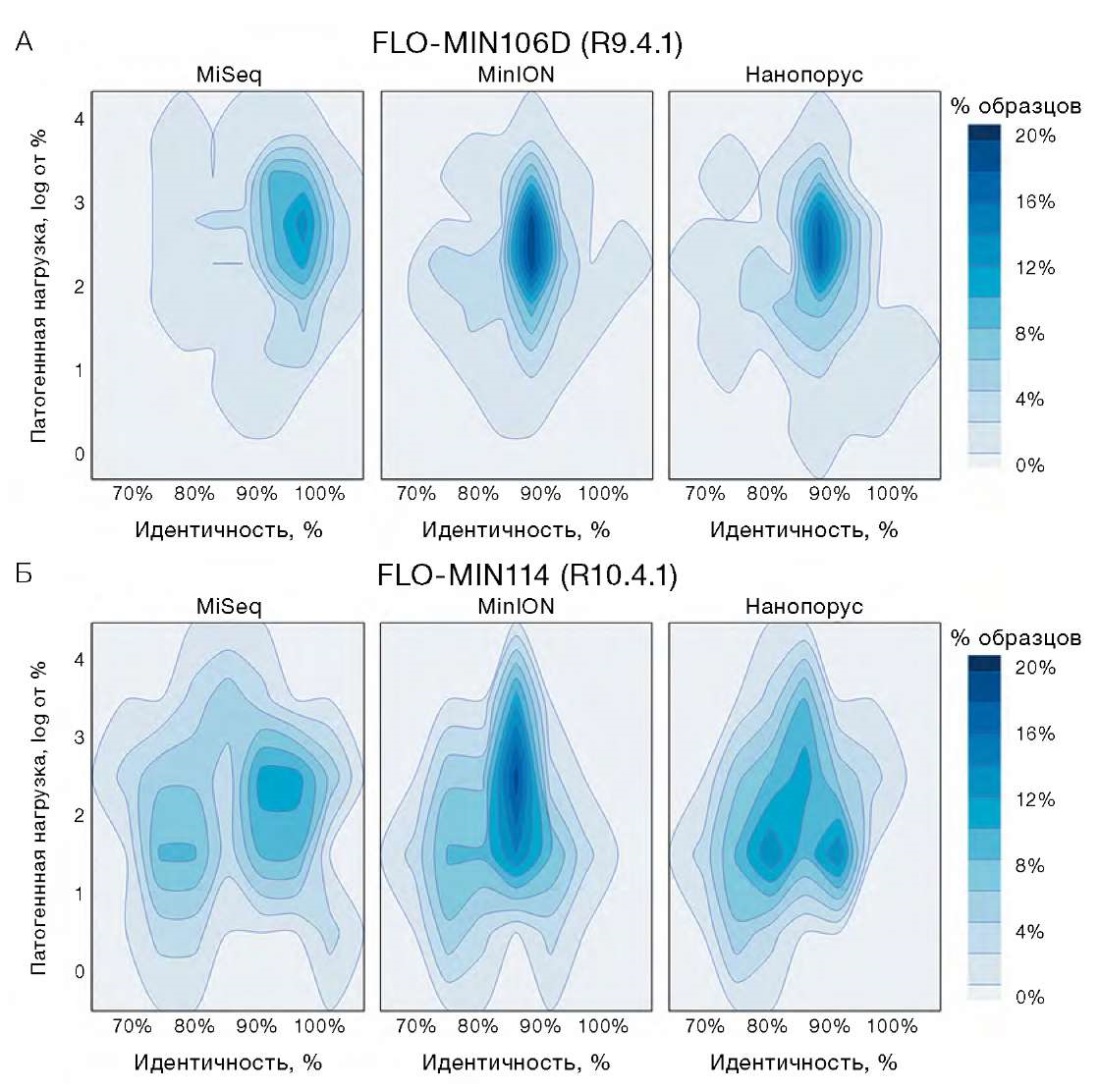
Рисунок подготовлен авторами
Рис. 5. Дисперсионное распределение патогенной нагрузки и процента идентичности для данных, полученных секвенаторами MiSeq, MinION и Нанопорус
Примечание: графики соответствуют данным, зарегистрированным при использовании проточных ячеек R9.4.1(A) и R10.4.1(Б).
ОБСУЖДЕНИЕ РЕЗУЛЬТАТОВ
Долгое время компания Oxford Nanopore Technologies (ONT) оставалась единственным разработчиком, предлагающим решения в области нанопорового секвенирования. Однако за последний год на рынок вышли несколько аналогичных платформ. В частности, сравнительно недавно были анонсированы китайские секвенаторы: QNome-3841 и QNome-3841hex от компании QitanTech, которые уже нашли применение в задачах судебно-медицинской генетики и исследованиях полных бактериальных геномов [27][28]; CycloneSEQ от компании MGI, показавший существенные результаты в сборке геномов de novo, а также в метагеномном и single-cell секвенировании согласно опубликованному разработчиками платформы исследованию [20]. На момент проведения исследования сравнительный анализ CycloneSEQ и Gnome с системами, такими как MinION, был невозможен ввиду недавнего анонса платформ. Нанопоровый секвенатор Нанопорус, представляющий собой аналог широко известного устройства MinION, был анонсирован в конце 2023 года и введен в коммерческое использование в начале 2024 года. На момент написания исследования авторам не удалось обнаружить в открытом доступе исследований, посвященных прямому сопоставлению характеристик данных приборов, в связи с чем наши выводы основаны исключительно на собственных экспериментальных данных.
В ходе тестирования секвенатора Нанопорус нарушений или отклонений в работе выявлено не было. Подтверждена совместимость прибора с оригинальными проточными ячейками двух последних версий, наборами для подготовки библиотек и программным обеспечением от компании Oxford Nanopore Technologies. Данные о скорости транслокации ДНК через нанопоры не выявили искажений сигнала, что указывает на отсутствие существенных отклонений от ожидаемых показателей качества передачи информации на управляющий компьютер. Показатели устойчивости поддержания температурного режима нанопоровыми секвенаторами свидетельствуют о минимальных колебаниях температуры, что важно для стабильной работы нанопор, обеспечения точности секвенирования, предотвращения деградации библиотеки и поддержания оптимальных условий функционирования ферментов, участвующих в процессе секвенирования. В рамках данного анализа установлено соответствие оборудования заявленным характеристикам и функциональным требованиям.
Обновленная проточная ячейка R10.4.1 значительно улучшает точность и стабильность секвенирования, что подтверждается увеличением средних значений параметра Qscore на обеих нанопоровых платформах по сравнению с предыдущей версией проточной ячейки. Существующие исследования в данной области подтверждают, что последняя версия проточных ячеек от компании ONT обеспечивает значительное улучшение точности и качества прочтений [21]. Согласно предоставленным авторами данным процент пар оснований с качеством прочтений Q15 для проточной ячейки R10.4.1 в шесть раз выше, чем для версии R9.4.1; в случае проведения бактериальной сборки генома авторам удалось собрать 97% генома посредством более ранней версии проточной ячейки, а в случае R10.4.1 это значение увеличилось до 98%. В нашем исследовании наблюдается аналогичное улучшение: доля прочтений с качеством ниже Q20 и Q30 значительно уменьшается; количество низкокачественных прочтений ниже Q20 снижается с 70 до 55%, а ниже Q30 — с 97,3 до 76%. Это указывает на повышение точности и надежности секвенирования с использованием новой химии V14 и проточных ячеек R10.4.1 от ONT.
Секвенирование второго поколения признано высокоточным и достоверным методом детекции нуклеиновых кислот инфекционных агентов благодаря высокому качеству чтения нуклеотидов, что позволяет классифицировать его как ведущую технологию среди альтернативных методов молекулярной диагностики [22]. Однако технология нанопорового секвенирования (ONT) предлагает ряд уникальных преимуществ, таких как компактность и мобильность, что особенно выделяется на фоне сложных оптических систем, необходимых для секвенирования второго поколения. Эти характеристики ONT позволяют эффективно использовать технологию в условиях ограниченных ресурсов и оперативных полевых исследований, что является важным фактором для внедрения данного метода в процессы эпидемиологического надзора.
Ранее проведенные исследования продемонстрировали потенциал ONT в качестве альтернативы высокопроизводительным платформам второго поколения. В частности, в рамках исследования, оценивающего возможности идентификации патогенов бактериальной природы с использованием секвенаторов MinION и Illumina (тип прибора второго поколения не уточняется), на обоих приборах успешно выполнена идентификация вида, серотипов, MLST профилей и подтипов шига-токсина в изолятах Escherichia coli (STEC) [23]. В ходе другого исследования, посвященного идентификации бактериального состава эталонного образца, содержащего 8 различных патогенов, установлено, что производительность классификации на уровне семейства и рода преобладала при использовании MinION. Тем не менее на уровне вида идентификация патогенов оказалась более точной на платформе MiSeq. Важно отметить, что для MinION использовалась проточная ячейка версии R9.4.1 [24]. Ряд других исследований, посвященных оценке эффективности применения разных поколений секвенирования для выявления вирусных патогенов, в частности представителей родов Alphavirus [25] и Adenovirus [26], также демонстрируют лишь незначительное преимущество платформ второго поколения по качеству идентификации в сравнении с результатами, полученными на нанопоровых секвенаторах. Наши данные подтверждают выводы, сделанные в вышеописанных исследованиях, демонстрируя высокую корреляцию между результатами детекции патогенов секвенаторами разных поколений. На приборах MinION и Нанопорус было идентифицировано 98,8 и 97,6% патогенов соответственно из числа инфекционных агентов, детектированных платформой MiSeq. Также установлено, что с увеличением патогенной нагрузки в образце наблюдается повышение уровня сходимости результатов секвенирования второго и третьего поколения. Особенно высокое соответствие отмечается между данными, полученными с платформ MinION и Нанопорус по мере превышения среднего значения титра нуклеиновых кислот патогенов в исследуемых образцах. В связи с этим ct MinION и Нанопорус можно считать взаимозаменяемыми в контексте задач по идентификации и количественной оценке патогенов.
В ходе исследования проведена оценка способности нанопоровых секвенаторов выявлять новые или малоизученные вирусные вариации. В выборке исследуемого контрольного материала присутствовали образцы, для которых предварительная идентификация патогенного состава с использованием платформы MiSeq выполнена с низкими показателями качества выравнивания, что может свидетельствовать об ограниченной представленности данных вариаций патогенов в существующих базах данных или на полное отсутствие информации о них. Анализ дисперсионного распределения и параметров качества таксономической идентификации демонстрировал значительное соответствие данных второго и третьего поколения. Это свидетельствует о способности нанопоровых секвенаторов MinION и Нанопорус выявлять ранее неизвестные или малоизученные вирусы на уровне, сопоставимом с высокопроизводительными платформами.
ЗАКЛЮЧЕНИЕ
Устройство Нанопорус продемонстрировало совместимость с проточными ячейками последних двух версий, наборами для подготовки библиотек и программным обеспечением компании Oxford Nanopore Technologies, что позволяет его интегрировать в существующие лабораторные процессы без необходимости значительных модификаций рабочих протоколов. Результаты сравнительного анализа подтвердили высокий уровень согласованности данных таксономической идентификации патогенного состава, полученных с использованием секвенаторов третьего поколения MinION и Нанопорус, с результатами, обеспечиваемыми платформой второго поколения MiSeq. На основании полученных данных ограничений для применения нанопоровых секвенаторов MinION и Нанопорус в лабораторных исследованиях по обнаружению патогенных микроорганизмов не выявлено.
Список литературы
1. Brown BL, Watson M, Minot SS, Rivera MC, Franklin RB. MinIONTM nanopore sequencing of environmental metagenomes: a synthetic approach. GigaScience. 2017;6(3):gix007. https://doi.org/10.1093/gigascience/gix007
2. Schmidt K, Mwaigwisya S, Crossman LC, Doumith M, Munroe D, Pires C. Identification of bacterial pathogens and antimicrobial resistance directly from clinical urines by nanopore-based metagenomic sequencing. J Antimicrob Chemother. 2017;72(1):104–14. https://doi.org/10.1093/jac/dkw397
3. Ciuffreda L, Rodríguez-Pérez H, Flores C. Nanopore sequencing and its application to the study of microbial communities. Comput Struct Biotechnol J. 2021;19:1497–511.https://doi.org/10.1016/j.csbj.2021.02.020
4. Jain M, Olsen HE, Paten B, Akeson M. The Oxford Nanopore MinION: delivery of nanopore sequencing to the genomics community. Genome Biol. 2016;17(1):239. https://doi.org/10.1186/s13059-016-1103-0
5. Leggett RM, Clark MD. A world of opportunities with nanopore sequencing. J Exp Bot. 28 2017;68(20):5419–29. https://doi.org/10.1093/jxb/erx289
6. Ahmed YW, Alemu BA, Bekele SA, Gizaw ST, Zerihun MF, Wabalo EK. Epigenetic tumor heterogeneity in the era of single-cell profiling with nanopore sequencing. Clin Epigenetics.2022;14(1):107. https://doi.org/10.1186/s13148-022-01323-6
7. Searle B, Müller M, Carell T, Kellett A. Third-Generation Sequencing of Epigenetic DNA. Angew Chem. 2023;135(14):e202215704. https://doi.org/10.1002/ange.202215704
8. Parker MT, Knop K, Sherwood AV, Schurch NJ, Mackinnon K, Gould PD. Nanopore direct RNA sequencing maps the complexity of Arabidopsis mRNA processing and m6A modification. Wan Y, Hardtke CS. eLife. 2020;9:e49658. https://doi.org/10.7554/eLife.49658
9. Quick J, Loman NJ, Duraffour S, Simpson JT, Severi E, Cowley L. Real-time, portable genome sequencing for Ebola surveillance. Nature. 2016;530(7589):228–32. https://doi.org/10.1038/nature16996
10. Ameur A, Kloosterman WP, Hestand MS. Single-Molecule Sequencing: Towards Clinical Applications. Trends Biotechnol. 2019;37(1):72–85. https://doi.org/10.1016/j.tibtech.2018.07.01
11. Sun X, Song L, Yang W, Zhang L, Liu M, Li X. Nanopore Sequencing and Its Clinical Applications. Methods Mol Biol Clifton NJ. 2020;2204:13–32. https://doi.org/10.1007/978-1-0716-0904-0_2
12. Wang M, Fu A, Hu B, Tong Y, Liu R, Liu Z. Nanopore Targeted Sequencing for the Accurate and Comprehensive Detection of SARS-CoV-2 and Other Respiratory Viruses. Small. 2020;16(32):2002169. https://doi.org/10.1002/smll.202002169
13. Chen Z, He X. Application of third-generation sequencing in cancer research. Med Rev. 2021;1:000010151520210013. https://doi.org/10.1515/mr-2021-0013
14. Lau BT, Almeda A, Schauer M, McNamara M, Bai X, Meng Q. Single-molecule methylation profiles of cell-free DNA in cancer with nanopore sequencing. Genome Med. 2023;15(1):33 https://doi.org/10.1186/s13073-023-01178-3
15. Faizuloev E, Mintaev R, Petrusha O, Marova A, Smirnova D, Ammour Y. New approach of genetic characterization of group A rotaviruses by the nanopore sequencing method. J Virol Methods. 2021;292:114114. https://doi.org/10.1016/j.jviromet.2021.114114
16. Tombácz D, Dörmő Á, Gulyás G, Csabai Z, Prazsák I, Kakuk B. High temporal resolution Nanopore sequencing dataset of SARS-CoV-2 and host cell RNAs. GigaScience. 2022;11:giac094. https://doi.org/10.1093/gigascience/giac094
17. Vacca D, Fiannaca A, Tramuto F, Cancila V, La Paglia L, Mazzucco W. Direct RNA Nanopore Sequencing of SARS-CoV-2 Extracted from Critical Material from Swabs. Life. 2022;12(1):69. https://doi.org/10.3390/life12010069
18. Gauthier NPG, Nelson C, Bonsall MB, Locher K, Charles M, MacDonald C. Nanopore metagenomic sequencing for detection and characterization of SARS-CoV-2 in clinical samples. Plos one. 2021;16(11):e0259712. https://doi.org/10.1371/journal.pone.0259712
19. Ben Chehida S, Filloux D, Fernandez E, Moubset O, Hoareau M, Julian C. Nanopore Sequencing Is a Credible Alternative to Recover Complete Genomes of Geminiviruses. Microorganisms. 2021;9(5):903. https://doi.org/10.3390/microorganisms9050903
20. Zhang, JY, Zhang, Y, Wang L, Guo F, Yun Q, Zeng T, Dong Y. A single-molecule nanopore sequencing platform. bioRxiv. 2024;08. https://doi.org/10.1101/2024.08.19.608720
21. Linde J, Brangsch H, Hölzer M, Thomas C, Elschner MC, Melzer F, et al. Comparison of Illumina and Oxford Nanopore Technology for genome analysis of Francisella tularensis, Bacillus anthracis, and Brucella suis. BMC Genomics.2023;24(1):258. https://doi.org/10.1186/s12864-023-09343-z
22. Satam H, Joshi K, Mangrolia U, Waghoo S, Zaidi G, Rawool S, et al. Next-Generation Sequencing Technology: Current Trends and Advancements. Biology. 2023;12(7):997. https://doi.org/10.3390/biology12070997
23. Greig DR, Jenkins C, Gharbia S, Dallman TJ. Comparison of single-nucleotide variants identified by Illumina and Oxford Nanopore technologies in the context of a potential outbreak of Shiga toxin–producing Escherichia coli. GigaScience. 2019;8(8):giz104. https://doi.org/10.1093/gigascience/giz104
24. Winand R, Bogaerts B, Hoffman S, Lefevre L, Delvoye M, Van Braekel J, et al.Targeting the 16S rRNA Gene for Bacterial Identification in Complex Mixed Samples: Comparative Evaluation of Second (Illumina) and Third (Oxford Nanopore Technologies) Generation Sequencing Technologies. Int J Mol Sci. 2020;21(1):298. https://doi.org/10.3390/ijms21010298
25. de Souza LM, de Oliveira ID, Sales FCS, da Costa AC, Campos KR, Abbud A, et al. Technical comparison of MinIon and Illumina technologies for genotyping Chikungunya virus in clinical samples. J Genet Eng Biotechnol. 2023;21:88. https://doi.org/10.1186/s43141-023-00536-3
26. Ye F, Han Y, Zhu J, Li P, Zhang Q, Lin Y, и др. First Identification of Human Adenovirus Subtype 21a in China With MinION and Illumina Sequencers. Front Genet. 2020. https://doi.org/10.3389/fgene.2020.00285
27. Peng K, Yin Y, Li Y, Qin S, Liu Y, Yang X, et al. QitanTech Nanopore Long-Read Sequencing Enables Rapid Resolution of Complete Genomes of Multi-Drug Resistant Pathogens. Frontiers in microbiology. 2022;13:778659. https://doi.org/10.3389/fmicb.2022.778659
28. Wang Z, Qin L, Liu J, Jiang L, Zou X, Chen X, et al. Forensic nanopore sequencing of microhaplotype markers using QitanTech’s QNome. Forensic science international. Genetics. 2022;57:102657. https://doi.org/10.1016/j.fsigen.2021.102657
Об авторах
Д. А. ГригорянРоссия
Григорян Диана Агароновна
Москва
И. Ф. Стеценко
Россия
Стеценко Иван Федорович
Москва
Б. С. Гуков
Россия
Гуков Борис Сергеевич
Москва
А. Д. Мацвай
Россия
Мацвай Алина Дмитриевна - канд. биол. наук
Москва
Г. А. Шипулин
Россия
Шипулин Герман Александрович - канд. мед. наук
Москва
Дополнительные файлы
Рецензия
Для цитирования:
Григорян Д.А., Стеценко И.Ф., Гуков Б.С., Мацвай А.Д., Шипулин Г.А. Сравнительный анализ нанопоровых секвенаторов MinION и Нанопорус в задаче идентификации нуклеиновых кислот патогенов. Медицина экстремальных ситуаций. 2025;27(1):64-73. https://doi.org/10.47183/mes.2024-242
For citation:
Grigoryan D.A., Stetsenko I.F., Gukov B.S., Matsvay A.D., Shipulin G.A. Comparative evaluation of MinION and Nanoporus nanopore sequencers in identification of pathogen nucleic acids. Extreme Medicine. 2025;27(1):64-73. https://doi.org/10.47183/mes.2024-242

















